生信方法实现基于功能基因的微生物群落预测模型
在《How Microbes Shape Their Communities? A Microbial Community Model Based on Functional Genes》一文中,作者利用生物信息分析实现了基于功能基因的微生物群落预测模型。
文章中生物信息分析部分主要包括宏基因组分析、统计学分析和FCP建模及预测,其中1)宏基因组分析采用的软件和我们平时常用的软件都不太一样,且很多软件包已不再维护,如果有涉及这方面的分析,建议大家使用目前的主流软件。2)统计学相关分析均在R语言环境下实现,主要包括GBM、MRT和CCREPE三个R包的使用。3)FCP 建模及预测是利用Matlab软件自带的FMINCON函数实现的,最后结合常规的统计学分析手段完成相关验证和CSS基因筛选。
宏基因组分析
在宏基因组分析流程中,首先对高通量测序下机的双端序列原始数据进行质量筛查,获取可用于下游分析的高质量数据集。随后对其进行宏基因组序列拼接组装,构建宏基因组Contigs序列集,并进行基因预测,获得非冗余蛋白序列集。最后,选择常用的数据库对蛋白序列进行功能注释,本研究采用是COG数据库。
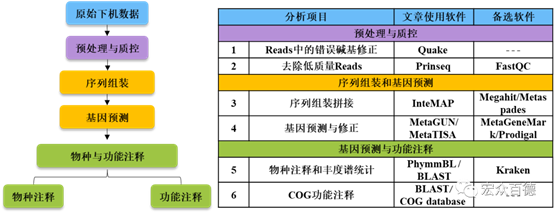
Step1: detect and correct errors in the raw data(Reads错误碱基修正)
Step2: filter out low quality reads(去除低质量Reads)
Step3: assemble these preprocessed reads into contigs(Reads组装)
Step4: predict protein coding genes(基因预测)
Step5: taxonomic classification(物种注释)
Step6: COG database annotation(COG功能注释)
统计学分析
文章利用MRT和GBM包分析解释复杂生态系统物种组成和环境因子之间的关系。使用CCREPE算法解析微生物群落中物种间相关丰度的关系,具体步骤如下。
1. CCREPE分析
Step1 安装和加载ccrepe包
Step2 构建测试数据集
Step3 运行ccrepe
2. MRT分析
Step1 安装和加载mvpart包
Step2 读入数据
Step3 运行mvpart
3. GBM分析
Step1 安装和加载gbm包
Step2 读入数据
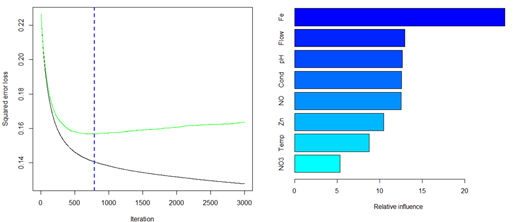
Step3 gbm建模
Step4 查看最佳迭代次数,如图左展示
Step5 看出最终结果,如图右展示
FCP 建模及预测
在matlab中,fmincon函数可以求解带约束的非线性多变量函数(Constrained nonlinear multivariable function)的最小值,即可以用来求解非线性规划问题,matlab中,非线性规划模型的写法如下:

FMINCON函数用法:
[x,fval]=fmincon(fun,x0,A,b,Aeq,beq,lb,ub,nonlcon,options)
x—返回值是决策向量x的取值,fval的返回值是目标函数f(x)的取值
fun—是用M文件定义的函数f(x),代表了(非)线性目标函数
x0 —是x的初始值
A,b,Aeq,beq—定义了线性约束 ,如果没有线性约束,则A=[],b=[],Aeq=[],beq=[]
lb和ub—变量x的下界和上界,如果下界和上界没有约束,则lb=[],ub=[],也可以写成lb的各分量都为 -inf, ub的各分量都为inf
nonlcon—用M文件定义的非线性向量函数约束
options—定义优化参数,不填写表示使用Matlab默认的参数设置
以上就是该文献生信分析方法体系的实现方法,欢迎感兴趣的朋友前来和我们交流、沟通。
